Crédit :Wiley
Qu'il s'agisse de révéler un agresseur avec des preuves ADN, diagnostiquer un agent pathogène, classer une découverte paléontologique, ou déterminer la paternité, la duplication des acides nucléiques (amplification) est indispensable. Dans la revue Angewandte Chemie , les scientifiques ont maintenant introduit un nouveau, très simple, méthode pourtant très sensible et fiable qui évite les étapes habituelles de chauffage et de refroidissement, ainsi que des instruments compliqués. Les réactifs peuvent être lyophilisés, permettant d'utiliser cette méthode universelle en dehors du laboratoire.
La méthode d'amplification la plus couramment utilisée est la réaction en chaîne par polymérase (PCR), qui est basé sur la répétition de plusieurs cycles thermiques dans des instruments spéciaux qui ont une forte demande de puissance. Il est difficile d'effectuer en dehors d'un laboratoire, au chevet d'un patient ou dans un endroit éloigné, par exemple. Les méthodes alternatives sans cycles thermiques sont souvent compliquées ou pas assez sensibles, nécessitent des réactifs coûteux, ou ne sont pas largement applicables.
Des chercheurs travaillant avec Bin-Cheng Yin et Bang-Ce Ye à l'East China University of Science &Technology, Shanghaï, Chine, ont maintenant développé un nouveau, méthode peu coûteuse :appelée réaction d'amplification basée sur Cas9n (Cas9nAR), il consiste en une seule étape en solution homogène, et se déroule à une température constante de 37 °C.
Dans cette approche, les chercheurs utilisent des composants du « système immunitaire » des bactéries. Lorsqu'une bactérie est infectée par un virus, par exemple, ils découpent le matériel génétique étranger en petits morceaux et les introduisent dans des zones spécifiques de leur propre génome. En cas d'infection ultérieure, les brins d'ARN de la bactérie « reconnaissent » ces séquences et dirigent des « ciseaux génétiques » spéciaux pour découper l'ADN étranger. Ces outils ont également été utilisés dans le génie génétique moderne.
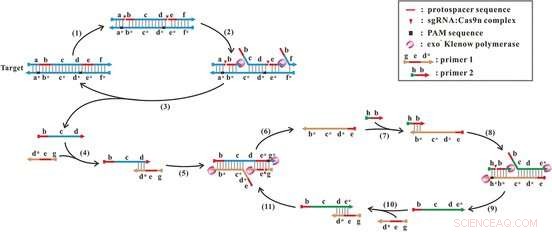
Yin, Vous, et leurs collègues ont modifié le ciseau génétique connu sous le nom de Cas9 afin qu'il ne coupe plus complètement l'ADN. Au lieu, il ne coupe qu'un seul brin, l'introduction d'un "nick". Ce type d'enzyme est appelé « nickase ». Comme dans le système bactérien, La nickase Cas9 se lie à un brin d'ARN, qui détermine l'emplacement du pseudo. Cet ARN peut être fabriqué pour qu'il reconnaisse une séquence d'ADN caractéristique d'un agent pathogène, par exemple. La nickase Cas9 coupe alors l'ADN immédiatement adjacent.
Pour la nouvelle technique, les chercheurs ont produit deux complexes d'ARN de nickase Cas9 différents, qui entaille l'ADN à deux endroits différents. Une polymérase couramment utilisée en PCR (exo(?) polymérase de Klenow) complète le brin coupé à partir de la première entaille, libérer l'ancien brin, pièce par pièce, jusqu'à ce qu'il atteigne le deuxième entaille. L'ADN nouvellement complété est entaillé à plusieurs reprises et complété par le complexe de nickase. Les courts brins simples que ce processus libère deviennent le point de départ d'une amplification supplémentaire dans un deuxième cycle. En plus du complexe de nickase et de la polymérase, les seules choses requises sont deux amorces appropriées comme points de départ pour les copies.
Des tests avec un fragment d'ADN génomique bactérien ont démontré que la séquence cible était précisément reconnue et amplifiée. Dans un volume de 20 l, il était possible de détecter une seule molécule. Les différences d'un seul nucléotide au sein d'un gène pourraient être détectées avec une grande spécificité.