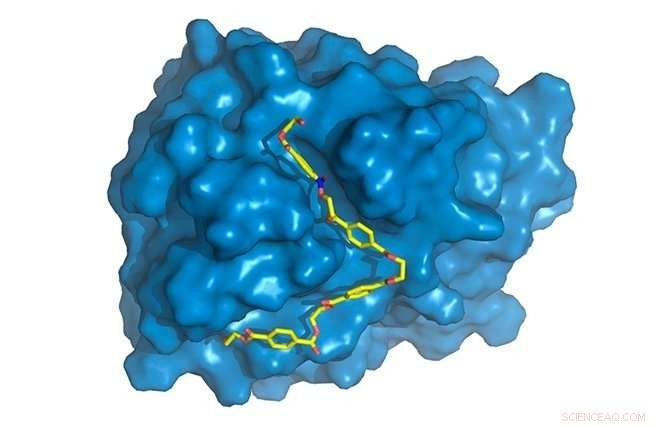
La PETase est représentée en bleu, avec chaîne PET (jaune) liée au site actif, où il sera dégradé. Crédit :Rodrigo Leandro Silveira
Depuis sa découverte, l'enzyme PETase a suscité un grand intérêt scientifique pour sa capacité à digérer le PET (polyéthylène téréphtalate).
Polymère utilisé principalement pour fabriquer des bouteilles de boissons (mais aussi des vêtements, les tapis, et autres produits), Le PET présente l'inconvénient de résister à la dégradation. Les bouteilles et autres objets en PET (polyéthylène téréphtalate) mettent au moins 800 ans à se biodégrader dans les décharges ou dans la mer. Entre 4,8 milliards et 12,7 milliards de kilogrammes de plastique sont déversés dans les océans chaque année.
Une étude dont les résultats ont été publiés récemment dans la revue Actes de l'Académie nationale des sciences ( PNAS ) a montré comment une équipe internationale de collaborateurs a réussi à augmenter la capacité de PETase à décomposer le plastique.
« Dans notre projet de recherche, nous avons caractérisé la structure tridimensionnelle de l'enzyme capable de digérer ce plastique, l'a conçu pour augmenter sa capacité de dégradation, et démontré qu'il agit également sur le polyéthylène-2, 5-furandicarboxylate (PEF), un substitut du PET fabriqué à partir de matières premières renouvelables, " a déclaré le co-auteur Rodrigo Leandro Silveira, stagiaire postdoctoral à l'Institut de chimie de l'Université de Campinas (IQ-UNICAMP).
Une bactérie qui survit en métabolisant le PET
L'intérêt pour la PETase est né en 2016, lorsqu'un groupe de chercheurs japonais dirigé par Shosuke Yoshida a identifié une nouvelle espèce de bactérie, Ideonella sakaiensis, qui peuvent se nourrir de PET en l'utilisant comme source de carbone et d'énergie. La bactérie reste le seul organisme connu avec cette capacité. Il pousse littéralement sur PET.
"En plus d'identifier I. sakaiensis, les scientifiques japonais ont découvert qu'il produit deux enzymes et les sécrète dans l'environnement, " expliqua Silveira. " L'une des enzymes sécrétées est la PETase. Parce qu'il a un certain degré de cristallinité, Le PET est un polymère très difficile à décomposer. Mais la PETase l'attaque et la décompose en petites unités d'acide mono(2-hydroxyéthyl) téréphtalique, ou MHET. Les unités de MHET sont ensuite converties en acide téréphtalique et absorbées et métabolisées par la bactérie."
I. Sakaiensis est le seul organisme vivant connu à utiliser une molécule synthétique fabriquée par l'homme pour survivre. Cela signifie que la bactérie est le résultat d'un processus évolutif très récent qui s'est déroulé au cours des dernières décennies. La bactérie s'est adaptée à un polymère développé au début des années 40 et n'a commencé à être utilisé à l'échelle industrielle que dans les années 70. La PETase est la clé pour comprendre comment.
"PETase fait le plus dur, qui décompose la structure cristalline et dépolymérise le PET en MHET, " a déclaré le chercheur financé par la FAPESP. " Le travail effectué par la deuxième enzyme, qui transforme le MHET en acide téréphtalique, est beaucoup plus simple, car son substrat est constitué de monomères auxquels l'enzyme peut accéder facilement car ils sont dispersés dans le milieu réactionnel. Pour cette raison, la recherche s'est concentrée sur la PETase."
L'enzyme modifiée se lie mieux au polymère
L'étape suivante consistait à étudier la PETase en détail, la contribution apportée par le nouveau projet de recherche. « Nous nous sommes concentrés sur la recherche de ce qui donne à la PETase la capacité de faire quelque chose que d'autres enzymes ne peuvent pas faire de manière très efficace. Nous avons commencé par caractériser la structure 3D de cette protéine, " expliqua Silveira.
"Obtenir la structure 3-D, c'est découvrir le x, coordonnées y et z de chacun des milliers d'atomes qui composent la macromolécule. Nos collègues britanniques l'ont fait en utilisant une technique bien connue et largement utilisée appelée diffraction des rayons X disponible dans un laboratoire très similaire à Sirius, actuellement en construction à Campinas."
Une fois la structure 3D obtenue, les chercheurs ont commencé à comparer la PETase avec des protéines apparentées. Le parent le plus proche est une cutinase de la bactérie Thermobifida fusca qui dégrade la cutine, une sorte de vernis naturel que l'on trouve sur les feuilles des plantes. Certains micro-organismes pathogènes utilisent la cutinase pour briser la barrière de la cutine et les nutriments appropriés dans les feuilles.
"Nous avons trouvé des différences spécifiques dans la PETase par rapport à la cutinase dans la région de l'enzyme où se produisent les réactions chimiques, connu comme le site actif. PETase a un site actif plus ouvert, par exemple, " a déclaré Silveira. "Nous avons étudié les mouvements moléculaires de l'enzyme grâce à des simulations informatiques, la partie à laquelle j'ai le plus contribué. Alors que la structure cristalline, obtenu par diffraction des rayons X, fourni des informations statiques, les simulations nous ont fourni des informations dynamiques et nous ont permis de découvrir le rôle spécifique de chaque acide aminé dans le processus de dégradation du PET."
La physique des mouvements de la molécule résulte de l'attraction et de la répulsion électrostatique d'un grand nombre d'atomes et de la température. Des simulations informatiques ont permis aux chercheurs de mieux comprendre comment la PETase se lie et interagit avec la TEP.
"Nous avons découvert que la PETase et la cutinase ont deux acides aminés différents au site actif. Nous avons ensuite utilisé des procédures de biologie moléculaire pour produire des mutations dans la PETase dans le but de la convertir en cutinase, " a déclaré Silveira.
« Si nous pouvions faire cela, nous découvririons quels composants lui ont donné cette propriété unique de dégrader le PET. Cependant, à notre surprise, lorsque nous avons essayé de supprimer cette activité particulière de la PETase - en essayant de convertir la PETase en cutinase - nous avons produit une PETase encore plus active. Nous avons essayé de réduire son activité, et au lieu de cela, nous l'avons boosté."
Davantage de simulations informatiques ont été nécessaires pour comprendre pourquoi la PETase mutante était meilleure que la PETase d'origine. La modélisation et les simulations ont clairement montré que les altérations produites dans la PETase d'origine facilitaient la liaison de l'enzyme au substrat. Cette liaison dépend à la fois de la géométrie, avec deux molécules s'assemblant comme une clé et un trou de serrure, et sur les facteurs thermodynamiques impliqués dans les interactions entre les différents composants de l'enzyme et du polymère. La manière élégante de décrire cela est que la PETase modifiée a une "plus grande affinité" pour le substrat.
En termes d'obtention d'une enzyme capable de digérer des tonnes de déchets plastiques, l'étude a été un grand succès, mais pourquoi PETase est PETase reste un mystère.