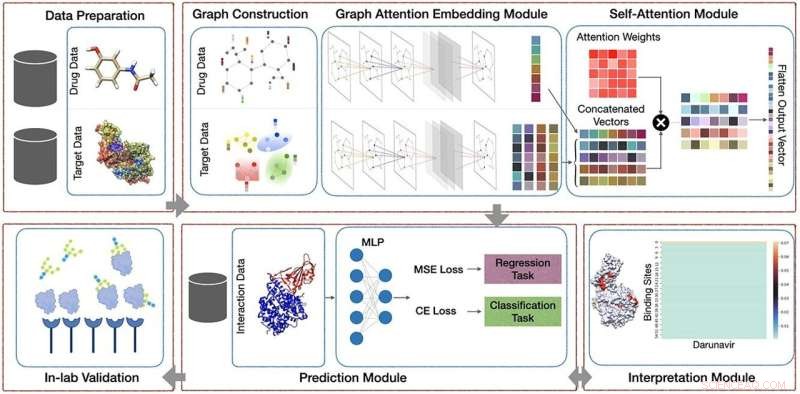
Notre cadre proposé comprend cinq modules principaux :(1) module de prétraitement qui consiste à trouver les sites de liaison des protéines; (2) Module d'apprentissage en profondeur AttentionSiteDTI, où nous construisons des représentations graphiques des sites de liaison SMILE et protéines des ligands, et nous créons un réseau neuronal convolutif de graphes armé d'un mécanisme de mise en commun de l'attention pour extraire les incorporations apprenables à partir de graphes, ainsi qu'un auto- mécanisme d'attention pour apprendre la relation entre les ligands et les sites de liaison des protéines ; (3) Module de prédiction pour prédire une interaction inconnue dans une paire médicament-cible, qui peut traiter à la fois des tâches de classification et de régression ; (4) Module d'interprétation pour mieux comprendre quels sites de liaison d'une protéine cible sont les plus susceptibles de se lier à un ligand donné. (5) Validations en laboratoire, où nous comparons nos résultats prédits par calcul avec les interactions médicament-cible observées expérimentalement (mesurées) en laboratoire pour tester et valider le potentiel pratique de notre modèle proposé. Crédit :Briefings en bioinformatique (2022). DOI :10.1093/bib/bbac272
Développer des médicaments vitaux peut prendre des milliards de dollars et des décennies de temps, mais les chercheurs de l'Université de Floride centrale visent à accélérer ce processus avec un nouveau processus de dépistage des médicaments basé sur l'intelligence artificielle qu'ils ont développé.
En utilisant une méthode qui modélise les interactions entre les médicaments et les protéines cibles à l'aide de techniques de traitement du langage naturel, les chercheurs ont atteint une précision allant jusqu'à 97 % dans l'identification de médicaments candidats prometteurs. Les résultats ont été publiés récemment dans la revue Briefings in Bioinformatics .
La technique représente les interactions médicament-protéine à l'aide de mots pour chaque site de liaison protéique et utilise l'apprentissage en profondeur pour extraire les caractéristiques qui régissent les interactions complexes entre les deux.
"Avec l'IA devenant plus disponible, c'est devenu quelque chose auquel l'IA peut s'attaquer", déclare le co-auteur de l'étude, Ozlem Garibay, professeur adjoint au Département de génie industriel et de systèmes de gestion de l'UCF. "Vous pouvez essayer de nombreuses variantes de protéines et d'interactions médicamenteuses et découvrir lesquelles sont les plus susceptibles de se lier ou non."
Le modèle qu'ils ont développé, connu sous le nom d'AttentionSiteDTI, est le premier à pouvoir être interprété à l'aide du langage des sites de liaison aux protéines.
Ce travail est important car il aidera les concepteurs de médicaments à identifier les sites critiques de liaison aux protéines ainsi que leurs propriétés fonctionnelles, ce qui est essentiel pour déterminer si un médicament sera efficace.
Les chercheurs ont réussi cet exploit en concevant un mécanisme d'auto-attention qui permet au modèle d'apprendre quelles parties de la protéine interagissent avec les composés médicamenteux, tout en obtenant des performances de prédiction de pointe.
La capacité d'auto-attention du mécanisme fonctionne en se concentrant sélectivement sur les parties les plus pertinentes de la protéine.
Les chercheurs ont validé leur modèle à l'aide d'expériences en laboratoire qui ont mesuré les interactions de liaison entre les composés et les protéines, puis ont comparé les résultats avec ceux que leur modèle avait prédits par calcul. Comme les médicaments pour traiter le COVID sont toujours intéressants, les expériences comprenaient également le test et la validation de composés médicamenteux qui se lieraient à une protéine de pointe du virus SARS-CoV2.
Selon Garibay, la grande concordance entre les résultats de laboratoire et les prédictions informatiques illustre le potentiel d'AttentionSiteDTI pour présélectionner des composés médicamenteux potentiellement efficaces et accélérer l'exploration de nouveaux médicaments et la réaffectation de ceux existants.
"Cette recherche à fort impact n'a été possible que grâce à une collaboration interdisciplinaire entre l'ingénierie des matériaux et l'IA/ML et les informaticiens pour faire face à la découverte liée au COVID", déclare Sudipta Seal, co-auteur de l'étude et président du Département de science et d'ingénierie des matériaux de l'UCF.
Mehdi Yazdani-Jahromi, doctorant au Collège d'ingénierie et d'informatique de l'UCF et auteur principal de l'étude, affirme que les travaux introduisent une nouvelle direction dans le dépistage préalable des médicaments.
"Cela permet aux chercheurs d'utiliser l'IA pour identifier les médicaments avec plus de précision afin de répondre rapidement aux nouvelles maladies", a déclaré Yazdani-Jahromi. "Cette méthode permet également aux chercheurs d'identifier le meilleur site de liaison d'une protéine virale sur lequel se concentrer dans la conception de médicaments."
"La prochaine étape de nos recherches consistera à concevoir de nouveaux médicaments utilisant la puissance de l'IA", déclare-t-il. "Cela peut naturellement être la prochaine étape pour se préparer à une pandémie." Les chercheurs identifient de nouveaux médicaments à l'aide de prédictions d'apprentissage en profondeur interprétables