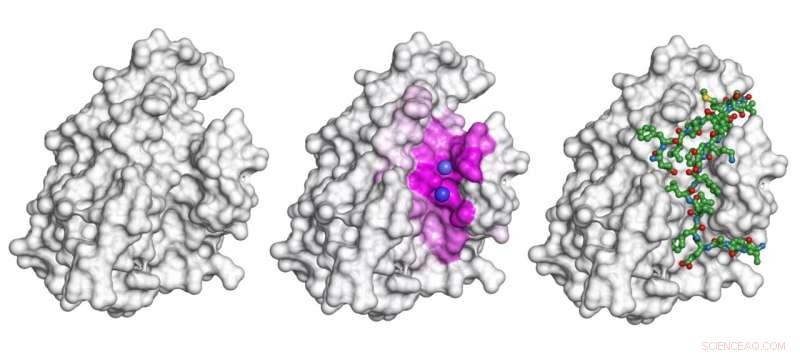
La forme grise est une protéine. Pour le scénario de cette protéine se liant au peptide montré comme un modèle de bâton et de balle verdâtre sur la droite, le modèle présenté dans l'étude met en évidence la surface impliquée dans l'interaction (la zone rose au milieu) et prédit les sites de liaison exacts (sphères violettes). Crédit :Igor Kozlovskii et Petr Popov / Skoltech
Deux chercheurs de Skoltech ont présenté un modèle de réseau neuronal très efficace qui utilise des données sur la structure des protéines pour prédire lesquelles de leurs parties interagissent avec d'autres molécules biologiques appelées peptides. Sachant cela est utile pour développer des médicaments à base de peptides, qui peuvent affecter les interactions protéine-protéine au sein des cellules de manière ciblée et non toxique, régulation d'un large éventail de processus cellulaires. L'étude est sortie dans le Journal of Chemical Information and Modelling .
Les protéines sont la machinerie des cellules, se déplacer, s'engager les uns avec les autres, et exécuter toutes sortes d'opérations. Les pharmacologues ont toujours été intrigués par la perspective de bricoler les interactions entre les protéines. Pourtant, ils semblaient être hors limites en tant que cible potentielle de médicaments :les molécules thérapeutiques plus grosses, appelés produits biologiques, ne pouvait pas pénétrer dans la cellule pour agir sur les protéines, tandis que les agents à petites molécules se sont souvent révélés incapables d'une telle action.
Peptides, qui médiatisent ou régulent naturellement environ 40 % des processus cellulaires, occupent un terrain d'entente prometteur et offrent des perspectives de médicaments ciblant les interactions protéine-protéine. Les peptides offrent le meilleur des deux mondes :comme les petites molécules, ils peuvent pénétrer la membrane cellulaire pour atteindre réellement leurs cibles, et ils présentent également une faible toxicité, ainsi qu'une affinité et une spécificité élevées (action forte et ciblée) - les caractéristiques des produits biologiques.
Pour concevoir des médicaments à base de peptides, les pharmacologues doivent connaître les sites dits de liaison pour toute cible protéique donnée. C'est-à-dire, les taches sur la protéine qui peuvent se lier à un peptide. Plus ces sites sont connus, plus il y a d'opportunités pour la conception de médicaments.
Les chercheurs peuvent identifier des sites de liaison expérimentalement, par exemple, utilisant la cristallographie aux rayons X, qui révèle la structure 3D des protéines cristallisées en étudiant comment elles diffractent les rayons X. Mais cela coûte très cher à faire pour une longue liste de molécules, et les méthodes de calcul offrent une alternative plus rapide et moins chère. Certains d'entre eux s'appuient sur des techniques d'apprentissage automatique, et à mesure que plus de données sur les structures des complexes protéine-peptide sont accumulées, ces méthodes deviennent plus puissantes et fournissent des prédictions de site de liaison toujours meilleures.
Dans leur article du 22 juillet dans le Journal of Chemical Information and Modelling , Doctorat Skoltech l'étudiant Igor Kozlovskii et le professeur assistant Petr Popov du groupe iMolecule ont présenté une méthode de calcul appelée BiteNetPp, qui exploite la puissance des réseaux de neurones convolutifs 3D pour détecter les sites de liaison protéine-peptide. Dans BiteNetPp, une structure protéique connue est alimentée à un réseau neuronal, qui met ensuite en évidence les sites de liaison peptidique suspectés, et génère un ensemble de coordonnées 3D putatives, ainsi que les scores de probabilité associés.
Petr Popov commente l'approche de la détection de sites de liaison en tant que reconnaissance d'image, introduit à l'origine dans l'article précédent de l'équipe et repris dans l'étude rapportée dans cet article :« Tout comme les réseaux de neurones peuvent être entraînés à reconnaître, dire, piétons ou cyclistes sur des photos 2D ordinaires, nous considérons la détection du site de liaison comme la détection d'un type particulier d'objet dans une image. La différence est que nous utilisons des données de structure atomique 3D comme entrées, donc le modèle fonctionne sur 'voxels, " un analogue tridimensionnel de pixels."
Le modèle nouvellement présenté s'appuie en fait sur celui de l'article précédent. "C'est ce qu'on appelle l'adaptation de domaine. BiteNetPp est le premier modèle à avoir été affiné sur un ensemble de données protéine-peptide après avoir été initialement formé sur des données protéine-petite molécule, " explique Popov. " Vous pouvez imaginer cela comme la formation d'un modèle pour identifier les endroits où les cyclistes ont tendance à s'arrêter dans la rue, mais vous commencez avec des données sur l'endroit où les piétons ont tendance à s'arrêter, et ce n'est qu'ensuite que vous étendez votre domaine aux cyclistes. Plutôt que de repartir de zéro, vous recyclez le modèle, anticipant que les « sites de liaison » pour les cyclistes pourraient partager certaines similitudes avec ceux qui attirent les piétons :vous savez, stands de glaces, feux de circulation, ce genre de chose."
Les créateurs du modèle ont démontré que BiteNetPp surpasse systématiquement les méthodes de pointe existantes en comparant leurs prédictions pour les sites de liaison protéine-peptide connus par des observations expérimentales. Surtout, le nouveau modèle prend moins d'une seconde pour analyser une seule structure protéique, ce qui le rend bien adapté aux études à grande échelle. Il existe des milliers d'interactions protéine-protéine potentiellement ciblables par des médicaments à base de peptides, les méthodes informatiques doivent donc être suffisamment rapides pour rendre leur criblage réalisable dans un contexte pharmacologique.