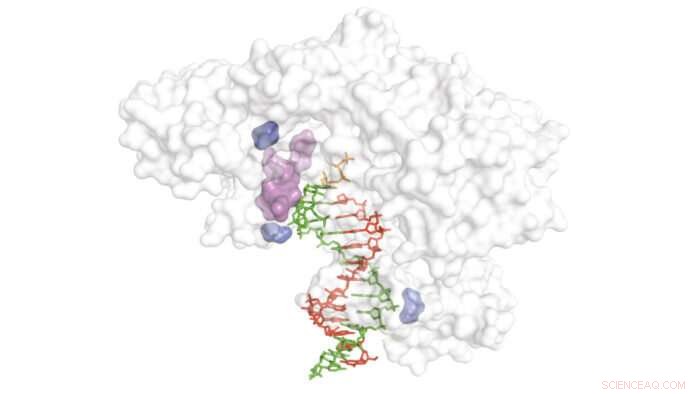
Diagramme montrant la structure de la transcriptase inverse (blanc) avec brin d'ADNc naissant (rouge), Modèle XNA (vert) et régions critiques pour la transcription inverse des modèles XNA (bleu et violet). Crédit :MRC Laboratoire de biologie moléculaire
En plus d'être requis pour certaines des techniques de base de la biologie moléculaire, les enzymes de la transcriptase inverse (RT) ont joué un rôle clé dans la génétique synthétique en permettant la synthèse, réplication, et l'évolution des acides xénonucléiques (XNA). Cependant, pour la plupart des chimies XNA, aucune enzyme RT n'est disponible ou les enzymes existantes ont une faible activité. le groupe de Philipp Holliger, dans la division PNAC de la LMB, ont développé une nouvelle méthode d'évolution dirigée pour améliorer l'activité RT pour toute chimie des acides nucléiques et découvert un nouveau groupe d'enzymes RT optimales.
Les XNA sont des polymères génétiques comme l'ADN ou l'ARN, mais avec des anneaux de sucre altérés, socles, ou des épines dorsales. Malgré ces chimies différentes, ils sont toujours capables de stocker et de transmettre des informations génétiques et peuvent effectuer des fonctions enzymatiques tout comme les enzymes à ARN, également connu sous le nom de ribozymes. Ils peuvent également fonctionner comme des aptamères et se lier à des protéines avec une spécificité et une affinité élevées, tout comme les anticorps. Ces fonctions et les propriétés variées résultant de leurs différentes chimies signifient que les XNA pourraient avoir un large éventail d'applications en biotechnologie et en médecine. Cependant, la génération d'aptamères XNA et de XNAzymes a été limitée par le manque d'enzymes RT de haute fidélité.
Qu'est-ce que la transcription inverse ?
La première étape du dogme dit central de la biologie moléculaire est la transcription de l'ADN pour produire de l'ARN. L'ARN peut également être rétrotranscrit pour produire de l'ADN et les enzymes RT synthétiques permettent d'accéder aux XNA.
En permettant aux scientifiques de convertir l'ARN en ADN, Les enzymes RT permettent aux scientifiques d'étudier plus facilement quels gènes sont transcrits à l'intérieur des cellules, et donc quels gènes sont " activés, " grâce à des techniques de base telles que la RT-PCR et le RNAseq. Parallèlement aux applications en recherche, cette capacité est également utilisée pour les tests médicaux, comme pour tester la présence d'ARN viral, y compris dans les tests COVID-19.
Pour remédier au manque d'enzymes RT haute fidélité, Gillian Houlihan et d'autres du groupe de Philipp ont développé une nouvelle technique d'évolution dirigée qui a conduit à la découverte d'un nouveau groupe d'enzymes RT optimales capables de décoder les informations génétiques de manière plus précise et plus efficace. Surtout, cette nouvelle méthode est compatible avec toute chimie des acides nucléiques et leur découverte comprend de nouvelles enzymes RT pour les chimies XNA pour lesquelles aucune enzyme RT n'existait auparavant. Parmi les nouvelles enzymes RT figurent les premières enzymes capables de relire activement lors de la transcription inverse XNA, amélioration de la précision.
Les enzymes ARN RT haute fidélité auront des applications immédiates dans la recherche et la biotechnologie car elles fourniront une précision de séquençage améliorée lors de l'analyse des ARN cellulaires ou viraux. L'amélioration de l'activité XNA RT contribuera probablement au développement de nouveaux aptamères XNA qui pourraient être utiles dans le diagnostic et la thérapeutique d'un large éventail de maladies. A titre d'exemple précis, ce travail comprend la première enzyme RT pour la chimie XNA utilisée dans l'oligo-médicament antisens nusinersen qui a obtenu l'approbation de la FDA et de l'EMA pour le traitement de l'amyotrophie spinale. La découverte de cette enzyme RT ouvre la possibilité de quantifier le niveau et la demi-vie de ce médicament chez les patients, ce qui pourrait faciliter le traitement.