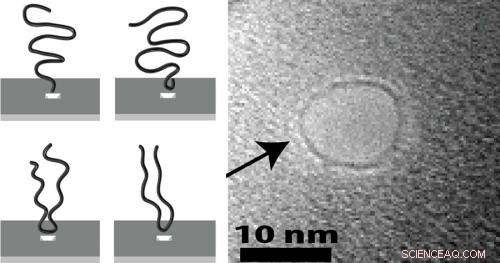
Lorsqu'un brin d'ADN est capturé et tiré à travers un nanopore, il est beaucoup plus probable qu'il commence le voyage à l'une de ses extrémités (en haut à gauche) plutôt que d'être saisi quelque part au milieu et tiré dans une configuration pliée. Crédit :Stein lab/Brown University
(Phys.org)—Dans les années 1960, Le lauréat du prix Nobel Pierre-Gilles de Gennes a postulé qu'un jour les chercheurs pourraient tester ses théories sur les réseaux de polymères en observant des molécules uniques. Les chercheurs de Brown ont observé des molécules uniques d'ADN attirées à travers des nanopores par un courant électrique et ont compris pourquoi elles voyageaient le plus souvent la tête la première.
Si vous voulez comprendre un roman, cela aide à commencer par le début plutôt que d'essayer de reprendre l'intrigue quelque part au milieu. Il en va de même pour l'analyse d'un brin d'ADN. La meilleure façon de lui donner un sens est de le regarder tête-bêche.
Heureusement, selon une nouvelle étude menée par des physiciens de l'Université Brown, Les molécules d'ADN ont une tendance commode à coopérer.
La recherche, publié dans la revue Lettres d'examen physique , examine la dynamique de la façon dont les molécules d'ADN sont capturées par les nanopores à l'état solide, de minuscules trous qui pourraient bientôt aider à séquencer l'ADN à la vitesse de l'éclair. L'étude a révélé que lorsqu'un brin d'ADN est capturé et tiré à travers un nanopore, il est beaucoup plus probable de commencer le voyage à l'une de ses extrémités, plutôt que d'être saisi quelque part au milieu et tiré dans une configuration pliée.
"Nous pensons qu'il s'agit d'une avancée importante pour comprendre comment les molécules d'ADN interagissent avec ces nanopores, " a déclaré Derek Stein, professeur adjoint de physique à Brown, qui a effectué la recherche avec les étudiants diplômés Mirna Mihovilivic et Nick Haggerty. "Si vous voulez faire du séquençage ou une autre analyse, vous voulez que la molécule traverse le pore tête-bêche."
Les recherches sur le séquençage de l'ADN avec des nanopores ont commencé il y a un peu plus de 15 ans. Le concept est assez simple. Un petit trou, quelques milliardièmes de mètre de diamètre, est fourré dans une barrière séparant deux piscines d'eau salée. Un courant électrique est appliqué à travers le trou, qui attire occasionnellement une molécule d'ADN flottant dans l'eau. Quand cela arrive, la molécule est fouettée à travers le pore en une fraction de seconde. Les scientifiques peuvent alors utiliser des capteurs sur le pore ou d'autres moyens pour identifier les bases nucléotidiques, les éléments constitutifs du code génétique.
La technologie évolue rapidement, et les premiers dispositifs de séquençage nanopore devraient être sur le marché très prochainement. Mais il reste des questions fondamentales sur le comportement des molécules au moment où elles sont capturées et avant.
"Ce que les molécules faisaient avant d'être capturées était un mystère et une question de spéculation, ", a déclaré Stein. "Et nous aimerions savoir parce que si vous essayez de concevoir quelque chose pour contrôler cette molécule - pour qu'elle fasse ce que vous voulez qu'elle fasse - vous devez savoir ce qu'elle fait."
Pour savoir ce que font ces molécules, les chercheurs ont soigneusement suivi plus de 1, 000 instances d'une molécule passant à travers un nanopore. Le courant électrique à travers le pore fournit un signal de la façon dont la molécule est passée. Les molécules qui passent par le milieu doivent d'abord être repliées pour pouvoir passer. Cette configuration pliée prend plus de place dans le pore et bloque une plus grande partie du courant. Donc, en regardant les différences dans le courant, Stein et son équipe ont pu compter combien de molécules ont traversé la tête la première et combien ont commencé quelque part au milieu.
L'étude a révélé que les molécules sont plusieurs fois plus susceptibles d'être capturées à ou très près d'une extrémité qu'à tout autre point le long de la molécule.
"Ce que nous avons découvert, c'est que les fins sont des endroits spéciaux, " dit Stein. " Le milieu est différent d'une fin, et cela a une conséquence sur la probabilité qu'une molécule commence son voyage par la fin ou le milieu."
Toujours de la place pour Jell-O
Comme il s'avère, il y a une vieille théorie qui explique assez bien ces nouveaux résultats expérimentaux. C'est la théorie de Jell-O.
Jell-O est un réseau de polymères, une masse de brins de polymère ondulés qui s'attachent les uns aux autres à des jonctions aléatoires. Les brins ondulés sont la raison pour laquelle Jell-O est un jiggly, semi-solide. La façon dont les brins de polymère se connectent les uns aux autres n'est pas sans rappeler la façon dont un brin d'ADN se connecte à un nanopore à l'instant où il est capturé. Dans l'eau, Les molécules d'ADN sont mélangées dans des gribouillis aléatoires un peu comme les molécules de gélatine dans Jell-O.
"Il existe une théorie puissante qui décrit de combien de manières les polymères de Jell-O peuvent s'arranger et s'attacher, ", a déclaré Stein. "Cela s'avère parfaitement applicable au problème de l'endroit où ces molécules d'ADN sont capturées par un nanopore."
Appliqué à l'ADN, la théorie Jell-O prédit que si vous deviez compter toutes les configurations possibles d'un brin d'ADN au moment de la capture, vous constaterez qu'il y a plus de configurations dans lesquelles il est capturé par sa fin, par rapport à d'autres points le long du brin. C'est un peu comme les chances d'obtenir une paire au poker par rapport aux chances d'obtenir un brelan. Vous avez plus de chances d'obtenir une paire simplement parce qu'il y a plus de paires dans le deck qu'il n'y a de triples.
Cette mesure de toutes les configurations possibles - une mesure de ce que les physiciens appellent l'entropie de la molécule - est tout ce qui est nécessaire pour expliquer pourquoi l'ADN a tendance à aller la tête en premier. Certains scientifiques avaient émis l'hypothèse que les brins seraient peut-être moins susceptibles de passer par le milieu, car les plier en deux nécessiterait de l'énergie supplémentaire. Mais cette énergie de pliage semble ne pas avoir d'importance du tout. Comme le dit Stein, "Le nombre de façons dont une molécule peut se retrouver avec sa tête collée dans le pore est tout simplement plus grand que le nombre de façons dont elle peut se trouver avec le milieu touchant le pore."
Ces théories des réseaux polymères existent en fait depuis un certain temps. Ils ont été proposés pour la première fois par le regretté prix Nobel Pierre-Gilles de Gennes dans les années 1960, et Bertrand Duplantier ont fait des avancées décisives dans les années 1980. Mihovilivic, L'étudiant diplômé de Stein et l'auteur principal de cette étude, dit que c'est en fait l'un des premiers tests de laboratoire de ces théories.
"Ils ne pouvaient pas être testés jusqu'à présent, quand nous pouvons réellement faire des mesures de molécule unique, " dit-elle. " [De Gennes] a postulé qu'un jour il serait possible de tester cela. Je pense qu'il aurait été très excité de voir cela se produire."